library(tidyverse)
library(janitor)clana: Estatística Aplicada à Hidrometria
Aplicação de modelos de regressão múltipla para solução de dados faltantes em séries históricas de dados hidrométricos.
Introdução
Este trabalho investiga a aplicabilidade de modelos de regressão múltipla no preenchimento de dados faltantes nas séries históricas de dados hidrométricos da Agência Nacional de Águas (ANA).
A ANA é uma entidade do governo brasileiro responsável pela realização de estudos, pesquisas, elaboração e fiscalização de normas reguladoras relacionadas aos recursos hídricos e ao saneamento básico (BRASIL, 2000).
A ANA disponibiliza dados através de um sistema público, o Sistema Nacional de Informações sobre Recursos Hídricos (SNIRH), onde é possível acessar medições de níveis fluviais, vazões, chuvas, clima, qualidade da água e sedimentos. As medições são coletadas por diferentes instituições e empresas ligadas à Rede Hidrometeorológica Nacional, coordenada pela ANA. Em 2018, a rede contava com 4641 pontos de monitoramento (“Portal HidroWeb”, 2023).
O principal conjunto de dados utilizado foi uma série histórica referente à estação de coleta de dados Jacareí (Código 58110002), localizada no Rio Paraíba do Sul e operada pelo Serviço Geológico do Brasil. A série consiste de 962 observações ao longo dos anos 2000 a 2023.
A filtragem dos dados disponibilizados pelo termo “Jacareí” retornou um total de 33 resultados, tabulados abaixo por seus prefixos:
| Nº | Categoria | Arquivos |
|---|---|---|
| 1 | chuvas | 12 |
| 2 | cotas | 5 |
| 3 | curvadescarga | 2 |
| 4 | PerfilTransversal | 2 |
| 5 | qualagua | 3 |
| 6 | ResumoDescarga | 4 |
| 7 | sedimentos | 1 |
| 8 | vazoes | 4 |
| Σ | Total | 33 |
Em sua maior parte, tratam-se de dados de chuvas e cotas. O termo cotas refere-se aos níveis de água dos rios (ANA, 2021).
As observações são distribuídas em 78 colunas, 62 delas para cada um dos 31 dias do mês e 31 informações de status para o dia respectivo:
Cotas:
Cota01Cota02(…)Cota30Cota31Status:
Cota01StatusCota02Status(…)Cota30StatusCota31Status
Antes destas 62 colunas, as primeiras 16 são:
EstacaoCodigoNivelConsistenciaDataHoraMediaDiariaTipoMedicaoCotasMaximaMinimaMediaDiaMaximaDiaMinimaMaximaStatusMinimaStatusMediaStatusMediaAnualMediaAnualStatus
O significado de algumas destas variáveis é dado no topo de cada arquivo disponibilizado pelo sistema Hidroweb:
NivelConsistencia: 1 = Bruto, 2 = Consistido
MediaDiaria: 0 = Não, 1 = Sim
TipoMedicaoCotas: 1 = Escala, 2 = Linígrafo, 3 = Data Logger
Status: 0 = Branco, 1 = Real, 2 = Estimado, 3 = Duvidoso, 4 = Régua Seca
Esta informação, que aparece antes dos dados estruturados, foi removida nos arquivos utilizados para análise neste trabalho, já que dificulta a leitura por ferramentas computacionais. O arquivo original foi preservado e acompanha os conjuntos processados.
Este relatório foi elaborado utilizando R como ferramenta principal, uma linguagem de programação voltada à estatística. Para publicação, foi utilizado também o sistema de cadernos Quarto, parte do mesmo ecossistema.
Parte dos conceitos utilizados foi desenvolvida paralelamente no trabalho clana, um estudo sobre Estruturas de Dados onde um outro conjunto da agência foi utilizado para o desenvolvimento de uma aplicação capaz de identificar e preencher lacunas usando modelos de regressão linear simples.
Visão geral
| Objetivo | Demonstrar a aplicabilidade de algoritmos de regressão linear múltipla na solução de dados faltantes em séries históricas de dados hidrométricos da Agência Nacional de Águas |
| Problema | Determinar programaticamente quais variáveis possuem correlações fortes |
| Métrica | Coeficiente de correlação |
| Público | Pessoas pesquisadoras, estudantes e profissionais da área de hidrometria, geometeorologia e gestão de recursos hídricos |
| Dados | cotas_C_58110002.csv |
Conjunto de dados utilizado:
AGÊNCIA NACIONAL DE ÁGUAS. cotas_C_58110002. Brasília, 2018. Disponível em: https://www.snirh.gov.br/hidroweb/. Acesso em: 29 jun. 2023
Resultados da filtragem para Jacareí
| Código | Estação | Tipo de dado |
|---|---|---|
58110001 |
JACAREÍ - RÉGUA DA MARGEM | Fluviométrica |
58110002 |
JACAREÍ | Fluviométrica |
58096000 |
UHE SANTA BRANCA JUSANTE | Fluviométrica |
58110000 |
UHE SANTA BRANCA JACAREÍ | Fluviométrica |
58044000 |
BAIRRO RIO COMPRIDO | Fluviométrica |
58138000 |
BAIRRO REMEDINHO | Fluviométrica |
58110010 |
JACAREÍ | Fluviométrica |
58138500 |
PTE. ACESSO RES. JAQUARI (próx. Brahma) | Fluviométrica |
O coeficiente de correlação, aqui obtido programaticamente para encontrar relações fortes entre as diferentes variáveis, foi utilizado como a métrica norteadora deste estudo através de uma matriz de correlação.
Carregamento
Nesta etapa, os dados foram carregados e preparados para o processamento.
O arquivo de dados original é disponibilizado no formato csv, com vírgulas separando casas decimais e ponto e vírgula separando as colunas.
Serão utilizadas as bibliotecas tidyverse e janitor para limpar e fazer o processamento inicial dos dados:
O termo cotas foi adotado para referir-se ao conjunto de dados no código.
cotas <- read_delim("dados/cotas_C_58110002_headless.csv", delim=";")Warning: One or more parsing issues, call `problems()` on your data frame for details,
e.g.:
dat <- vroom(...)
problems(dat)Rows: 962 Columns: 78
── Column specification ────────────────────────────────────────────────────────
Delimiter: ";"
chr (3): Data, Hora, Cota31Status
dbl (75): EstacaoCodigo, NivelConsistencia, MediaDiaria, TipoMedicaoCotas, M...
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Limpeza
Pela saída da importação é possível ver que há problemas. A mesma mensagem indica como listá-los:
problems(cotas)row <int> | col <int> | expected <chr> | actual <chr> | |
|---|---|---|---|---|
| 2 | 79 | 78 columns | 79 columns | |
| 3 | 79 | 78 columns | 79 columns | |
| 4 | 79 | 78 columns | 79 columns | |
| 5 | 79 | 78 columns | 79 columns | |
| 6 | 79 | 78 columns | 79 columns | |
| 7 | 79 | 78 columns | 79 columns | |
| 8 | 79 | 78 columns | 79 columns | |
| 9 | 79 | 78 columns | 79 columns | |
| 10 | 79 | 78 columns | 79 columns | |
| 11 | 79 | 78 columns | 79 columns |
Parece que cada linha é terminada com um caracter ; extra.
Será portanto necessário limpar este caracter da última coluna e alterar o tipo para numérico:
cotas$Cota31Status <- as.numeric(gsub(";$", "", cotas$Cota31Status))O conjunto carregado agora possui uma estrutura uniforme, ainda que com muitas lacunas:
Código
cotas |>
filter(
grepl("01/04/2022", Data)
)EstacaoCodigo <dbl> | NivelConsistencia <dbl> | Data <chr> | Hora <chr> | |
|---|---|---|---|---|
| 58110002 | 1 | 01/04/2022 | 01/01/1900 07:00:00 | |
| 58110002 | 1 | 01/04/2022 | 01/01/1900 17:00:00 | |
| 58110002 | 1 | 01/04/2022 | NA |
Acima, uma filtragem demonstrativa das observações referentes ao mês de abril de 2022.
Cabeçalhos
Os cabeçalhos do arquivo original foram processados automaticamente, e não possuem caracteres especiais ou espaços:
colnames(cotas) [1] "EstacaoCodigo" "NivelConsistencia" "Data"
[4] "Hora" "MediaDiaria" "TipoMedicaoCotas"
[7] "Maxima" "Minima" "Media"
[10] "DiaMaxima" "DiaMinima" "MaximaStatus"
[13] "MinimaStatus" "MediaStatus" "MediaAnual"
[16] "MediaAnualStatus" "Cota01" "Cota02"
[19] "Cota03" "Cota04" "Cota05"
[22] "Cota06" "Cota07" "Cota08"
[25] "Cota09" "Cota10" "Cota11"
[28] "Cota12" "Cota13" "Cota14"
[31] "Cota15" "Cota16" "Cota17"
[34] "Cota18" "Cota19" "Cota20"
[37] "Cota21" "Cota22" "Cota23"
[40] "Cota24" "Cota25" "Cota26"
[43] "Cota27" "Cota28" "Cota29"
[46] "Cota30" "Cota31" "Cota01Status"
[49] "Cota02Status" "Cota03Status" "Cota04Status"
[52] "Cota05Status" "Cota06Status" "Cota07Status"
[55] "Cota08Status" "Cota09Status" "Cota10Status"
[58] "Cota11Status" "Cota12Status" "Cota13Status"
[61] "Cota14Status" "Cota15Status" "Cota16Status"
[64] "Cota17Status" "Cota18Status" "Cota19Status"
[67] "Cota20Status" "Cota21Status" "Cota22Status"
[70] "Cota23Status" "Cota24Status" "Cota25Status"
[73] "Cota26Status" "Cota27Status" "Cota28Status"
[76] "Cota29Status" "Cota30Status" "Cota31Status" Não foi necessária qualquer limpeza e eles puderam ser mantidos como no original, o que trouxe facilidade na verificação cruzada com o conjunto original e ferramentas auxiliares tais como planilhas.
Análise
Abaixo obteve-se uma listagem de quantas lacunas há em cada coluna:
Código
# obtém a quantidade de entradas nulas em cada coluna
total_lacunas <- colSums(is.na(cotas))
# filtra colunas sem valores nulos
total_lacunas <- total_lacunas[total_lacunas > 0]
# ordena os resultados de forma decrescente
total_lacunas <- total_lacunas[order(total_lacunas, decreasing = TRUE)]
as_tibble(
enframe(total_lacunas, name = "column", value = "n"))column <chr> | n <dbl> | |||
|---|---|---|---|---|
| MediaAnual | 782 | |||
| Hora | 444 | |||
| Cota31 | 440 | |||
| Cota31Status | 151 | |||
| Cota30 | 145 | |||
| Cota29Status | 135 | |||
| Cota01Status | 134 | |||
| Cota02Status | 134 | |||
| Cota03Status | 134 | |||
| Cota04Status | 134 |
Correlação
Usando a função cor() é possível chegar a uma matriz de coeficientes de correlação entre cada variável:
Código
# TODO: Considerar o desvio padrão
library(dplyr)
library(Matrix)
matriz <- cotas |>
select_if(is.numeric) |>
cor(use = "pairwise.complete.obs") |>
as_tibble()
matriz |>
add_column(Coluna = colnames(cotas)
[sapply(cotas, is.numeric)], .before = 1)Coluna <chr> | EstacaoCodigo <dbl> | NivelConsistencia <dbl> | MediaDiaria <dbl> | |
|---|---|---|---|---|
| EstacaoCodigo | NA | NA | NA | |
| NivelConsistencia | NA | 1.0000000000 | 0.51821057 | |
| MediaDiaria | NA | 0.5182105745 | 1.00000000 | |
| TipoMedicaoCotas | NA | NA | NA | |
| Maxima | NA | -0.0694707626 | -0.04217493 | |
| Minima | NA | -0.0411037066 | -0.02901073 | |
| Media | NA | -0.0505854671 | -0.03485165 | |
| DiaMaxima | NA | 0.0504187583 | 0.03150562 | |
| DiaMinima | NA | 0.0005889856 | -0.00145271 | |
| MaximaStatus | NA | 0.8900153792 | 0.49668630 |
A matriz de correlação obtida acima usa um método de correlação em pares para a covariância, parâmetro que indica o grau de interdependência linear entre duas variáveis em relação às suas médias.
Com a opção pairwise.complete.obs, são considerados apenas os pares onde nenhum dos dois valores é nulo. Como desvantagem, ela pode reduzir demais o tamanho da amostra quando há uma quantidade grande de valores nulos.
Temos agora valores nulos apenas para as colunas e linhas onde todos os valores eram nulos originalmente. Para removê-los:
matriz <- remove_empty(matriz)A documentação do pacote stats, ao qual pertence a função cor() utilizada acima, explica especificamente sobre a opção de completação de lacunas em pares:
“[for
pairwise.complete.obs] the correlation or covariance between each pair of variables is computed using all complete pairs of observations on those variables. This can result in covariance or correlation matrices which are not positive semi-definite, as well as NA entries if there are no complete pairs for that pair of variables.” (R CORE TEAM, 2019)
Validação do instrumento
A documentação também menciona que esta opção, em tradução livre, “pode resultar em matrizes de covariância ou correlação que não são positivas semi-definidas”, o que tem implicações que prejudicam a aplicabilidade da matriz na análise estatística.
Para compreender melhor o conceito de uma matriz positiva semi-definida (PSD) e aferir o efeito da escolha do método pairwise.complete.obs, foram utilizados duas técnicas de decomposição para verificar se a matriz é ou não uma matriz PSD.
Método de decomposição de autovalores
Código
matriz_tipada <- as.matrix(matriz)
autovalores <- eigen(matriz_tipada)$values
if(all(autovalores >= 0)) {
print("A matriz é PSD")
} else {
print("A matriz não é PSD")
}[1] "A matriz não é PSD"Método de decomposição de Cholesky
Código
triangular_inferior <- tryCatch(chol(matriz), error = function(e) NULL)
if(is.null(triangular_inferior)) {
print("A matriz não é PSD")
} else {
if(all.equal(matriz, crossprod(triangular_inferior))) {
print("A matriz é PSD")
}
}[1] "A matriz não é PSD"Os resultados mostram que a matriz obtida não é positiva semi-definida. Testes com a opção na.or.complete também não resultaram em uma matriz PSA.
Observando a matriz de autovalores, temos:
eigen(matriz)$values [1] 3.304671e+01 2.677692e+01 3.976231e+00 1.868539e+00 1.084119e+00
[6] 8.337463e-01 8.183780e-01 6.740758e-01 5.365000e-01 3.227351e-01
[11] 3.085505e-01 2.171506e-01 1.817382e-01 1.622910e-01 1.322591e-01
[16] 1.272485e-01 1.165717e-01 8.765272e-02 8.596270e-02 6.983907e-02
[21] 6.669530e-02 6.525508e-02 5.276238e-02 5.167361e-02 4.577009e-02
[26] 4.220613e-02 4.029004e-02 3.450093e-02 3.342940e-02 2.778568e-02
[31] 2.680202e-02 2.403391e-02 2.206614e-02 1.898203e-02 1.804426e-02
[36] 1.680097e-02 1.493449e-02 1.384304e-02 1.293349e-02 1.227241e-02
[41] 1.090765e-02 1.013699e-02 8.931990e-03 8.324636e-03 8.039344e-03
[46] 7.093028e-03 6.553614e-03 6.112212e-03 5.389511e-03 4.489292e-03
[51] 4.464281e-03 3.555311e-03 3.313878e-03 2.388201e-03 2.022481e-03
[56] 1.866073e-03 8.053837e-04 4.753483e-16 3.391994e-16 1.514189e-16
[61] 4.503450e-17 -9.757525e-17 -1.097625e-16 -2.994173e-06 -3.754538e-05
[66] -2.829942e-03 -3.100618e-03 -5.936686e-03 -1.285535e-02 -3.209864e-02
[71] -4.439433e-02 -6.143459e-02Parece que os autovalores negativos estão todos após a posição 62.
Recorte
Foi realizado um recorte utilizando apenas os dados referentes às cotas de cada 31 dias:
Código
recorte <- cotas |>
select(Cota01:Cota31) |>
drop_na()
recorteCota01 <dbl> | Cota02 <dbl> | Cota03 <dbl> | Cota04 <dbl> | Cota05 <dbl> | Cota06 <dbl> | Cota07 <dbl> | Cota08 <dbl> | Cota09 <dbl> | Cota10 <dbl> | |
|---|---|---|---|---|---|---|---|---|---|---|
| 78 | 78 | 78 | 78 | 75 | 74 | 74 | 71 | 72 | 74 | |
| 72 | 71 | 70 | 68 | 55 | 57 | 60 | 74 | 71 | 70 | |
| 75 | 75 | 74 | 73 | 65 | 66 | 67 | 73 | 72 | 72 | |
| 119 | 119 | 119 | 113 | 106 | 105 | 105 | 106 | 106 | 106 | |
| 119 | 119 | 119 | 119 | 105 | 105 | 105 | 106 | 106 | 106 | |
| 119 | 119 | 119 | 106 | 106 | 105 | 105 | 105 | 106 | 106 | |
| 167 | 167 | 168 | 168 | 180 | 180 | 180 | 180 | 188 | 188 | |
| 167 | 167 | 168 | 168 | 180 | 180 | 180 | 180 | 188 | 188 | |
| 167 | 167 | 168 | 168 | 180 | 180 | 180 | 180 | 188 | 188 | |
| 158 | 158 | 158 | 158 | 158 | 162 | 162 | 162 | 162 | 162 |
Em seguida, o recorte foi limpo de colunas ou linhas contendo campos vazios.
Código
matriz_recorte <- recorte |>
select_if(is.numeric) |>
cor(use = "na.or.complete") |>
as_tibble()
matriz_recorteCota01 <dbl> | Cota02 <dbl> | Cota03 <dbl> | Cota04 <dbl> | Cota05 <dbl> | Cota06 <dbl> | Cota07 <dbl> | |
|---|---|---|---|---|---|---|---|
| 1.0000000 | 0.9843837 | 0.9749178 | 0.9666425 | 0.9558252 | 0.9559924 | 0.9448886 | |
| 0.9843837 | 1.0000000 | 0.9843187 | 0.9745932 | 0.9596469 | 0.9606027 | 0.9493034 | |
| 0.9749178 | 0.9843187 | 1.0000000 | 0.9892490 | 0.9636828 | 0.9600585 | 0.9505056 | |
| 0.9666425 | 0.9745932 | 0.9892490 | 1.0000000 | 0.9783869 | 0.9679394 | 0.9521808 | |
| 0.9558252 | 0.9596469 | 0.9636828 | 0.9783869 | 1.0000000 | 0.9876378 | 0.9661084 | |
| 0.9559924 | 0.9606027 | 0.9600585 | 0.9679394 | 0.9876378 | 1.0000000 | 0.9793599 | |
| 0.9448886 | 0.9493034 | 0.9505056 | 0.9521808 | 0.9661084 | 0.9793599 | 1.0000000 | |
| 0.9381880 | 0.9413465 | 0.9393227 | 0.9414484 | 0.9586847 | 0.9698278 | 0.9848786 | |
| 0.9336140 | 0.9330056 | 0.9281905 | 0.9301548 | 0.9498871 | 0.9638331 | 0.9775429 | |
| 0.9310289 | 0.9309882 | 0.9264741 | 0.9288885 | 0.9485362 | 0.9619393 | 0.9712373 |
A recontagem de valores nulos mostra que não restaram mais lacunas:
Código
# obtém a quantidade de entradas nulas em cada coluna
lacunas <- colSums(is.na(matriz_recorte))
# filtra colunas sem valores nulos
lacunas <- lacunas[lacunas > 0]
# ordena os resultados de forma decrescente
lacunas <- lacunas[order(lacunas, decreasing = TRUE)]
as_tibble(enframe(lacunas, name = "column", value = "n"))column <chr> | n <dbl> |
|---|
Método de decomposição de autovalores
O novo recorte foi testado com sucesso pela decomposição de autovalores:
Código
tryCatch( autovalores <- eigen(matriz_recorte)$values , error = function(e) NULL)
if(is.null(autovalores)) {
print("Erro:")
print(e)
} else {
if(all(autovalores >= 0)) {
print("A matriz é PSD")
} else {
print("A matriz não é PSD")
}
}[1] "A matriz é PSD"Embora com este recorte seja possível um resultado positivo usando a técnica de verificação pela decomposição de autovalores, o fato da matriz não ser simétrica torna impossível realizar a segunda etapa necessária ao teste de decomposição de Cholesky.
O teste abaixo verifica se a matriz é simétrica:
Código
matriz_recorte <- as.matrix(matriz_recorte)
if (isSymmetric(matriz_recorte)) {
print("A matriz é simétrica")
} else {
print("A matriz não é simétrica")
}[1] "A matriz não é simétrica"Uma solução para o problema é completar a forma simétrica da matriz pela sua transposta.
Método de decomposição de Cholesky com a matriz completada
Código
# obtém a matriz simétrica somando-a com a transposta e dividindo por 2
matriz_simetrica <- (matriz_recorte + t(matriz_recorte)) / 2
triangular_inferior <- tryCatch(chol(matriz_simetrica), error = function(e) NULL)
triangular_superior <- row(triangular_inferior) > col(triangular_inferior)
all(triangular_inferior[triangular_superior] == 0)[1] TRUEForam levantados ainda os seguintes testes adicionais, que também confirmaram a matriz como PSD:
Teste por decomposição em valores singulares
Código
decomposicao <- svd(matriz_recorte)
all(decomposicao$d >= 0)[1] TRUETeste pela forma quadrática
Código
n_colunas = ncol(matriz_recorte)
x <- sample(-9:9, n_colunas, replace = TRUE)
while (all(x == 0)) {
x <- sample(-9:9, n_colunas, replace = TRUE)
}
resultado <- TRUE
for (i in 1:n_colunas) {
if (t(x) %*% matriz_recorte %*% x < 0) {
resultado <- FALSE
break
}
}
resultado[1] TRUEAplicação
Utilizando esta região, podemos fitlrar o conjunto original para ver como ela é incluindo suas lacunas.
Código
cotas |>
select(Cota01:Cota31)Cota01 <dbl> | Cota02 <dbl> | Cota03 <dbl> | Cota04 <dbl> | Cota05 <dbl> | Cota06 <dbl> | Cota07 <dbl> | Cota08 <dbl> | Cota09 <dbl> | Cota10 <dbl> | |
|---|---|---|---|---|---|---|---|---|---|---|
| NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | |
| NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | |
| NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | |
| 85 | 82 | 82 | 83 | 84 | 82 | 81 | 82 | 83 | 84 | |
| 81 | 82 | 83 | 84 | 82 | 82 | 81 | 82 | 83 | 84 | |
| 88 | 82 | 81 | 82 | 85 | 82 | 81 | 82 | 83 | 84 | |
| 78 | 78 | 78 | 78 | 75 | 74 | 74 | 71 | 72 | 74 | |
| 72 | 71 | 70 | 68 | 55 | 57 | 60 | 74 | 71 | 70 | |
| 75 | 75 | 74 | 73 | 65 | 66 | 67 | 73 | 72 | 72 | |
| 119 | 119 | 119 | 113 | 106 | 105 | 105 | 106 | 106 | 106 |
Podemos encontrar diversas áreas onde o modelo desenvolvido pode ser testado. Por exemplo, a região abaixo:
Código
cotas |>
select(Cota05:Cota16) |>
slice(745:755)Cota05 <dbl> | Cota06 <dbl> | Cota07 <dbl> | Cota08 <dbl> | Cota09 <dbl> | Cota10 <dbl> | Cota11 <dbl> | Cota12 <dbl> | Cota13 <dbl> | Cota14 <dbl> | |
|---|---|---|---|---|---|---|---|---|---|---|
| 34 | 51 | 46 | 33 | 28 | 27 | 28 | 37 | 48 | 43 | |
| 34 | 28 | 28 | 28 | 26 | 24 | 23 | 23 | 23 | 22 | |
| 33 | 36 | NA | NA | NA | NA | NA | NA | 19 | 25 | |
| 37 | 35 | NA | NA | NA | NA | NA | 19 | 19 | 22 | |
| 35 | 36 | NA | NA | NA | NA | NA | NA | 19 | 24 | |
| 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 110 | |
| 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 113 | |
| 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 108 | 112 | |
| 65 | 74 | 74 | 73 | 74 | 72 | 68 | 67 | 67 | 67 | |
| 69 | 74 | 74 | 73 | 74 | 71 | 67 | 67 | 67 | 67 |
Esta região corresponde aos dias 7 a 12 de abril de 2022.
Na tabela abaixo, vemos a mesma matriz de correlação com uma coluna extra à frente que facilita verificar a correlação entre as observações mostradas no intervalo.
Código
matriz_df <- matriz_recorte |>
as.data.frame()
matriz_df |>
add_column(Colunas = colnames(matriz_recorte), .before = 1)Colunas <chr> | Cota01 <dbl> | Cota02 <dbl> | Cota03 <dbl> | Cota04 <dbl> | Cota05 <dbl> | Cota06 <dbl> | |
|---|---|---|---|---|---|---|---|
| Cota01 | 1.0000000 | 0.9843837 | 0.9749178 | 0.9666425 | 0.9558252 | 0.9559924 | |
| Cota02 | 0.9843837 | 1.0000000 | 0.9843187 | 0.9745932 | 0.9596469 | 0.9606027 | |
| Cota03 | 0.9749178 | 0.9843187 | 1.0000000 | 0.9892490 | 0.9636828 | 0.9600585 | |
| Cota04 | 0.9666425 | 0.9745932 | 0.9892490 | 1.0000000 | 0.9783869 | 0.9679394 | |
| Cota05 | 0.9558252 | 0.9596469 | 0.9636828 | 0.9783869 | 1.0000000 | 0.9876378 | |
| Cota06 | 0.9559924 | 0.9606027 | 0.9600585 | 0.9679394 | 0.9876378 | 1.0000000 | |
| Cota07 | 0.9448886 | 0.9493034 | 0.9505056 | 0.9521808 | 0.9661084 | 0.9793599 | |
| Cota08 | 0.9381880 | 0.9413465 | 0.9393227 | 0.9414484 | 0.9586847 | 0.9698278 | |
| Cota09 | 0.9336140 | 0.9330056 | 0.9281905 | 0.9301548 | 0.9498871 | 0.9638331 | |
| Cota10 | 0.9310289 | 0.9309882 | 0.9264741 | 0.9288885 | 0.9485362 | 0.9619393 |
Modelo demonstrativo
Usando a função nativa em R para criar modelos de regressão linear, podemos criar um modelo utilizando duas ou mais variáveis. Para a coluna 7, podemos usar as colunas completas mais próximas, 6 e 5, que possuem coeficientes de correlação maiores.
Código
model = lm(Cota07~Cota06 + Cota05, data = matriz_df)O modelo retornado contém diferentes parâmetros para esta correlação específica:
Call:
lm(formula = Cota07 ~ Cota06 + Cota05, data = matriz_df)
Residuals:
Min 1Q Median 3Q Max
-0.0233749 -0.0020224 0.0001333 0.0019238 0.0148345
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.09288 0.05660 1.641 0.112
Cota06 1.95404 0.21484 9.095 7.48e-10 ***
Cota05 -1.05726 0.17802 -5.939 2.16e-06 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.006388 on 28 degrees of freedom
Multiple R-squared: 0.8949, Adjusted R-squared: 0.8874
F-statistic: 119.2 on 2 and 28 DF, p-value: 2.01e-14É possível destacar, por exemplo, os coeficientes:
(Intercept) Cota06 Cota05
0.0928818 1.9540448 -1.0572619 E Cota07, Cota06 e Cota05:
[1] 0.8948865Código
library(ggplot2)
ggplot(matriz_df, aes(x = Cota07, y = Cota06)) +
geom_point() +
geom_smooth(method = "lm", formula = y ~ x, se = FALSE, color = "darkgreen") +
labs(x = "Cota 07", y = "Cota 06") +
annotate("text", x = max(matriz_df$Cota07), y = max(matriz_df$Cota06),
label = paste("r:", round(cor(matriz_df$Cota07, matriz_df$Cota06), 2)),
hjust = 1, vjust = 1)
Código
library(ggplot2)
ggplot(matriz_df, aes(x = Cota07, y = Cota01)) +
geom_point() +
geom_smooth(method = "lm", formula = y ~ x, se = FALSE, color = "darkgreen") +
labs(x = "Cota 07", y = "Cota 01") +
annotate("text", x = max(matriz_df$Cota07), y = max(matriz_df$Cota01),
label = paste("r:", round(cor(matriz_df$Cota07, matriz_df$Cota01), 2)),
hjust = 1, vjust = 1)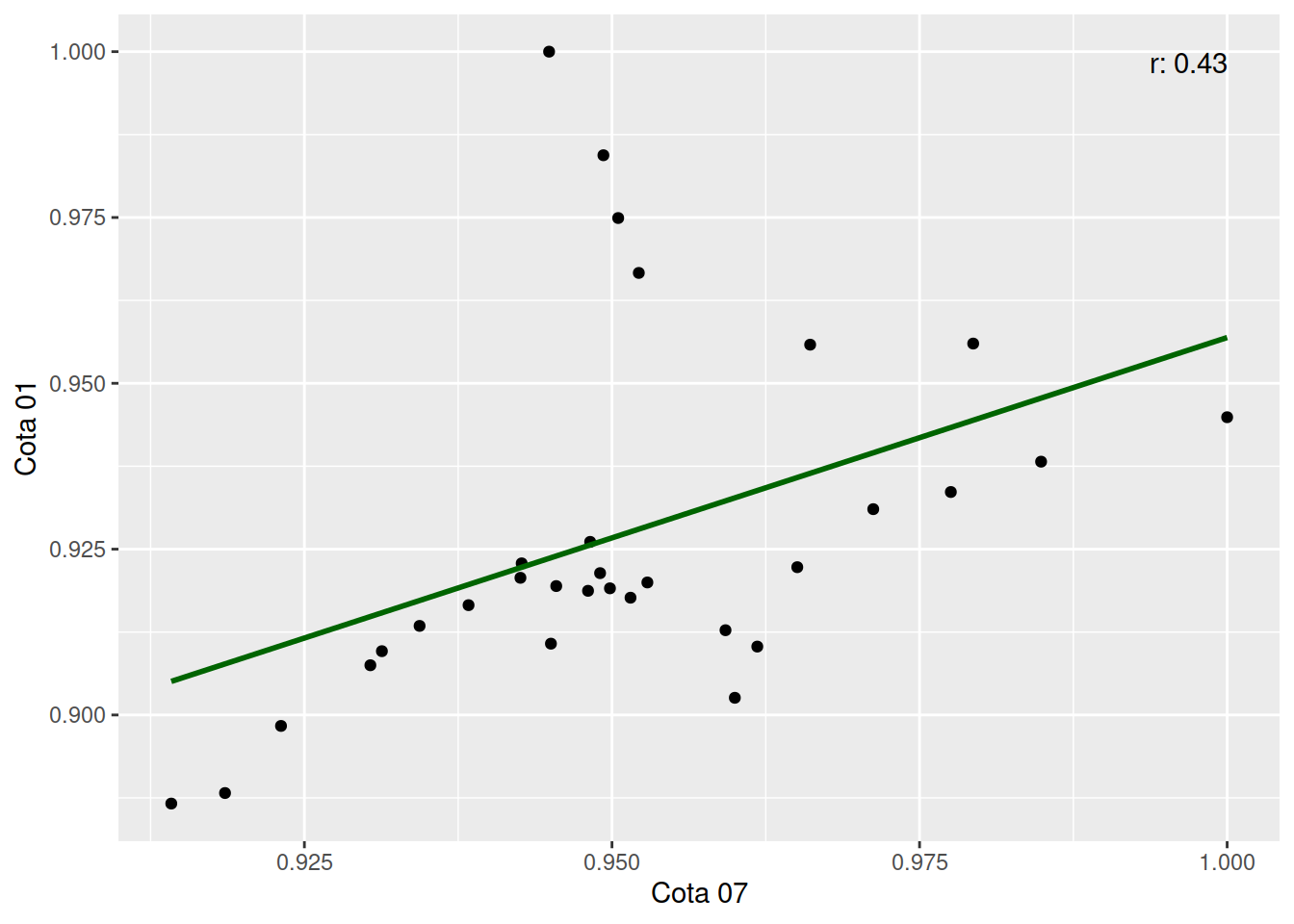
Código
library(ggplot2)
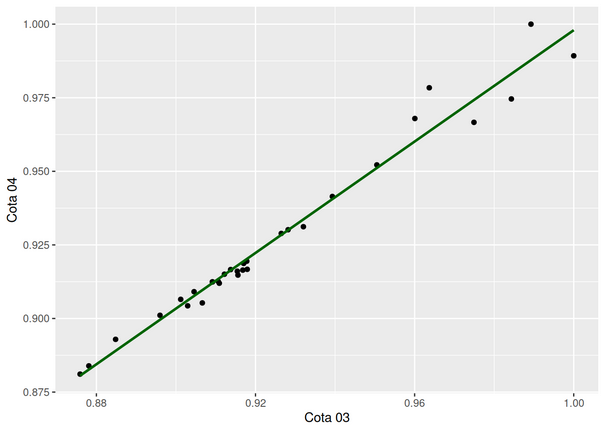
ggplot(matriz_df, aes(x = Cota03, y = Cota04)) +
geom_point() +
geom_smooth(method = "lm", formula = y ~ x, se = FALSE, color = "darkgreen") +
labs(x = "Cota 03", y = "Cota 04") +
annotate("text", x = max(matriz_df$Cota03), y = max(matriz_df$Cota04),
label = paste("r:", round(cor(matriz_df$Cota03, matriz_df$Cota04), 2)),
hjust = 1, vjust = 1)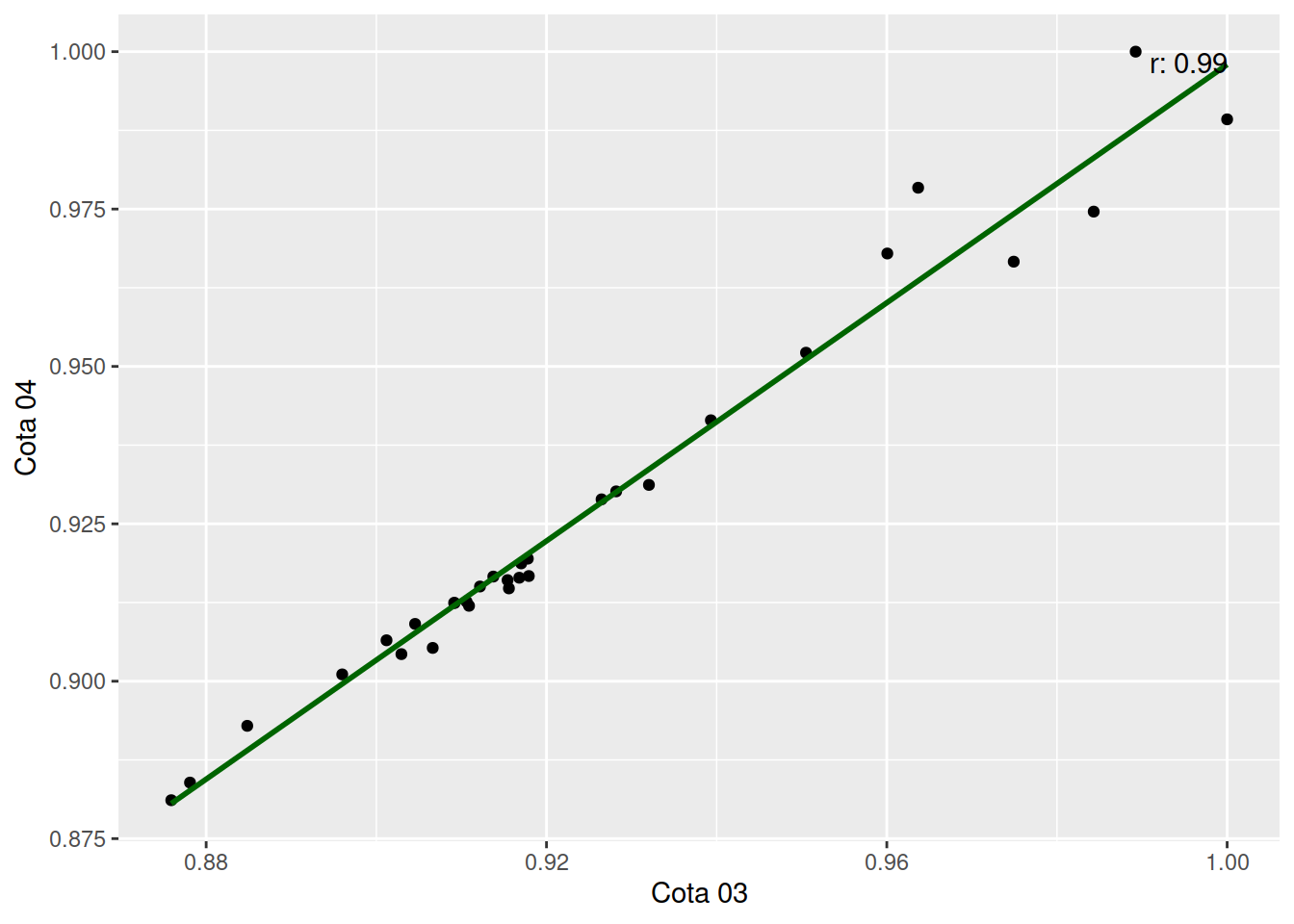
Conclusão
Para cenários onde os níveis dos rios não passam por mudanças bruscas, a regressão linear pode ser um instrumento eficaz na previsão de valores faltantes, em especial onde há dados em dias contíguos que podem ser usados na construção do modelo.
Como o estudo tomou como principal métrica os coeficientes de correlação, numericamente os dados mais sintéticos sobre o que foi observado estão na matriz de correlação obtida:
Cota01 <dbl> | Cota02 <dbl> | Cota03 <dbl> | Cota04 <dbl> | Cota05 <dbl> | Cota06 <dbl> | Cota07 <dbl> | |
|---|---|---|---|---|---|---|---|
| 1.0000000 | 0.9843837 | 0.9749178 | 0.9666425 | 0.9558252 | 0.9559924 | 0.9448886 | |
| 0.9843837 | 1.0000000 | 0.9843187 | 0.9745932 | 0.9596469 | 0.9606027 | 0.9493034 | |
| 0.9749178 | 0.9843187 | 1.0000000 | 0.9892490 | 0.9636828 | 0.9600585 | 0.9505056 | |
| 0.9666425 | 0.9745932 | 0.9892490 | 1.0000000 | 0.9783869 | 0.9679394 | 0.9521808 | |
| 0.9558252 | 0.9596469 | 0.9636828 | 0.9783869 | 1.0000000 | 0.9876378 | 0.9661084 | |
| 0.9559924 | 0.9606027 | 0.9600585 | 0.9679394 | 0.9876378 | 1.0000000 | 0.9793599 | |
| 0.9448886 | 0.9493034 | 0.9505056 | 0.9521808 | 0.9661084 | 0.9793599 | 1.0000000 | |
| 0.9381880 | 0.9413465 | 0.9393227 | 0.9414484 | 0.9586847 | 0.9698278 | 0.9848786 | |
| 0.9336140 | 0.9330056 | 0.9281905 | 0.9301548 | 0.9498871 | 0.9638331 | 0.9775429 | |
| 0.9310289 | 0.9309882 | 0.9264741 | 0.9288885 | 0.9485362 | 0.9619393 | 0.9712373 |
Os dados mostram uma tendência visível que varia de mais para menos conforme a variável se distancia da data em que o valor observado de fato ocorreu.
Embora neste recorte específico possamos perceber uma tendência relativamente linear, cabe ressaltar que diferentes fatores podem afetar as cotas, alguns linearmente relacionados e outros não, como a precipitação em pontos distantes que chegam aos rios por escoamento superficial e subterrâneo (CHOW et al., 1994 apud CAPOZZOLI et al., 2017).
As ferramentas utilizadas podem ser readaptadas para diferentes intervalos e tipos de conjuntos com poucas adaptações. Com isto a contribuição na criação de modelos documentáveis e transparentes também aparece como uma possibilidade interessante.
Bibliografia
AGÊNCIA NACIONAL DE ÁGUAS. Sistema Hidroweb - Séries históricas. Brasília, 2018. Disponível em: https://www.snirh.gov.br/hidroweb/. Acesso em: 29 jun. 2023
AGÊNCIA NACIONAL DE ÁGUAS. Levantamentos topobatimétricos e geodésicos aplicados na Rede Hidrometeorológica Nacional. Brasília, 2021. Disponível em: https://www.gov.br/ana/pt-br/assuntos/monitoramento-e-eventos-criticos/monitoramento-hidrologico/orientacoes-manuais/documentos/manual-de-nivelamento. Acesso em: 29 jun. 2023
GUZMÁN, N. G. Modelagem para estimativa de dados faltantes em série de dados meteorológicos. Dissertação (Mestrado em Modelagem Computacional) — Nova Friburgo: Universidade do Estado do Rio de Janeiro, 5 abr. 2018.
BRASIL. Poder Executivo. Lei Nº 9.984, de 17 de julho de 2000. Dispõe sobre a criação da Agência Nacional de Águas e Saneamento Básico. Brasília, DF: Poder Executivo, 2000. Disponível em: http://www.planalto.gov.br/ccivil_03/leis/l9984.htm
R CORE TEAM. R: A language and environment for statistical computing. Vienna, Áustria. R Foundation for Statistical Computing, 2018. Disponível em: https://www.R-project.org/. Acesso em: 29 jun. 2023
ALLAIRE, J. J. et al. Quarto. 23 jun. 2023. Disponível em: https://quarto.org/
R CORE TEAM. cor: Correlation, Variance and Covariance (Matrices). Disponível em: https://www.rdocumentation.org/packages/stats/versions/3.6.2/topics/cor. Acesso em: 1 jul. 2023.
CAPOZZOLI, C. R.; CARDOSO, A. DE O.; FERRAZ, S. E. T. Padrões de Variabilidade de Vazão de Rios nas Principais Bacias Brasileiras e Associação com Índices Climáticos. Revista Brasileira de Meteorologia, v. 32, p. 243–254, jun. 2017.
Recursos relacionados